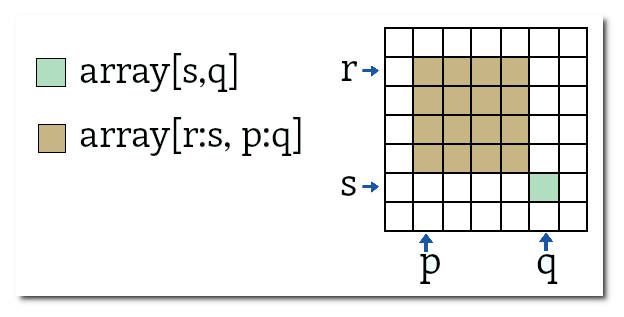Numpy, introdução

Numpy
NumPy é uma biblioteca do python especializada em computação científica e análise de dados. Ela é usada em diversos tipos de operações que envolvem operações matriciais. Além disso suas matrizes formam a base para outros pacotes, como o pandas e outras voltadas para o cálculo matemático e científico. Numpy foi primeiro lançado por Travis Oliphant em 2006 e tem sido mantido por um grande número de colabores desde então, sob licença BSD.
NumPy, com suas matrizes, apresenta algumas vantagens sobre o cálculo usual com objetos do python, como listas e tuplas. Essas operações são mais rápidas e flexíveis e foram construídas de forma a evitar a necessidade da realização de laços (loops ). Ela contém métodos voltados para operações da álgebra linear, geração de números aleatórios e transformadas de Fourier, além da interface voltada para a conexão com as linguagens C, C++ e FORTRAN.
Um exemplo rápido pode mostrar como as rotinas do numpy são mais eficientes que as de objetos list do python.
» # comparação de velocidades
» import numpy as np
» # um array do numpy
» array = np.arange(1_000_000)
» # uma lista usual do python
» lista = list(range(1_000_000))
» %time for _ in range(100): arr2 = array * 2
↳ CPU times: user 145 ms, sys: 7.93 ms, total: 152 ms
Wall time: 151 ms
» %time for _ in range(100): lista2 = [x * 2 for x in lista]
↳ CPU times: user 5.35 s, sys: 778 ms, total: 6.13 s
Wall time: 6.12 s
» # 5.35/.145 ≈ 37 × mais rápido
Wall time é o tempo total gasto pelo código para ser executado. CPU time é uma medida do tempo gasto pelo processador apenas quando esteve operando sobre a tarefa específica. Dependendo do cálculo feito o numpy pode ser mais de 100 vezes mais rápido que uma operação similar em puro python.
Instalação

Em geral o módulo está presente como pacote na maioria das distribuições de Python. Se necessária a sua instalação em separado pode ser feita. Numpy e pandas são instalados juntos com o Anaconda.
# No Linux (Ubuntu and Debian):
» sudo apt-get install python-numpy
# No Linux (Fedora)
» sudo yum install numpy scipy
# No Windows com Anaconda:
» conda install numpy
# após a instalação o módulo deve ser importado:
» import numpy as np
# O aliás np é opcional e de escolha do programador.
Ndarray
O objeto básico da biblioteca Numpy é o ndarray (N-dimensional array). Ndarrays são matrizes multidimensionais com número determinado de dimensões e elementos. Seus elementos pertencem todos a um único tipo, chamado de dtype (data-type ou tipo de dado). Cada dimensão é denominada por axis (eixo) e o número de eixos é o rank do objeto. Diferente das listas do python, ndarrays têm dimensões fixas, definidas em sua construção.
Um ndarray possui os seguintes atributos referentes ao seu tipo de dado, tamanho e ordem:
| Atributo |
descrição |
array.dtype |
tipo de dado armazenado. (Veja lista abaixo), |
array.ndim |
número de dimensões (que são eixos ou axis ); o mesmo que rank |
array.size |
número de elementos em cada eixo, |
array.shape |
(ou forma), tupla de N inteiros positivos com o comprimento de cada eixo. |
Um ndarray de 1 dimensão é um objeto similar a um vetor (rank = 1): arr1D = ([a0,a1,...,aN-1,]), arr1D.size = N, arr1D.ndim = 1, arr1D.shape = (N,).
Um ndarray de 2 dimensões é um objeto similar a uma matriz (rank = 2): se ela possui M linhas, cada uma com N elementos então arr2D.size = M × N , arr2D.ndim = 2, arr2D.shape = (M,N).
Um ndarray de 3 dimensões é uma coleção de matrizes (rank = 3): se ela possui K matrizes de M linhas, cada uma com N elementos então arr3D.size = K × M × N , arr3D.ndim = 3, arr3D.shape = (K, M, N).

Ndarrays de ordem superior são generalizações desse processo, acrescentados novos eixos.
Os eixos são numerados para diversas operações. Em 2 dimensões axis=0 são as linhas, axis=1 as colunas, e assim consecutivamente para ordens superiores.
Tipos, dtypes
Além dos tipos usuais do python, a importação de Numpy disponibiliza um conjunto extendido de tipos ou dtypes.
| dtype |
descrição |
bool |
booleano (true ou false) armazenado como um byte |
intX |
inteiro com sinal, X-bit (X=8,16,32, 64) |
uintX |
inteiro sem sinal, X-bit (X=8,16,32, 64) |
intc |
idêntical ao int C (em geral int32 ou int64) |
intp |
inteiro usado para indexação (como C size_t; em geral int32 ou int64) |
float_ |
o mesmo que float64 |
float16 |
meia precisão float: sign bit, 5-bit exponente, 10-bit mantissa |
float32 |
simple precisão float: sign bit, 8-bit exponente, 23-bit mantissa |
float64 |
dupla precisão float: sign bit, 11-bit exponente, 52-bit mantissa |
complex_ |
o mesmo que complex128 |
complex64 |
complexo, representado por dois 32-bit floats (parte real e imaginária) |
complex128 |
complexo, representado por dois 64-bit floats (parte real e imaginária) |

Construção de um array
Um array do NumPy pode ser criado passando-se uma lista para construtor np.array(lista).
» import numpy as np
» lista = [123,234,345]
» arr = np.array(lista)
» arr
↳ array([123, 234, 345])
# o objeto criado é um ndarray do numpy
» type(arr)
↳ numpy.ndarray
» arr.dtype
↳ dtype('int64')
» arr.ndim
↳ 1
» arr.shape
↳ (3,)
» arr.size
↳ 3
» # uma lista de listas
» lista2 = [[123,234,345],
[456,567,678],
[789,890,901]]
» arr2 = np.array(lista2)
» arr2
↳ array([[123, 234, 345],
[456, 567, 678],
[789, 890, 901]])
» arr2.ndim
↳ 2
» arr2.size
↳ 9
» arr2.shape
↳ (3, 3)
» # lista de listas de listas
» lista3 =[ [ [1,2],[2,3] ], [ [3,4],[4,5] ], [ [1,2],[2,3] ], [ [3,4],[4,5] ] ]
» arr3 = np.array(lista3)
» # o resultado é: 4 matrizes de 2 x 2 elementos
» arr3
↳ array([[[1, 2],
[2, 3]],
[[3, 4],
[4, 5]],
[[1, 2],
[2, 3]],
[[3, 4],
[4, 5]]])
» arr3.ndim
↳ 3
» arr3.shape
↳ (4, 2, 2)
» arr3.size
↳ 16
» # a 1ª matriz
» arr3[0]
↳ array([[1, 2],
[2, 3]])
» # a 2ª linha da 1ª matriz
» arr3[0,1]
↳ array([2, 3])
» # o 1º elemento da 2ª linha da 1ª matriz
» arr3[0,1,0]
↳ 2
# todos os arrays criados tem o mesmo dtype
» arr3.dtype
↳ dtype('int64')
Arrays podem ser de outros tipos, como um array de strings. No entanto devem ser homogêneos (todos os elementos do mesmo tipo). Uma tentativa de criar um array como em stArr2 causa uma tentativa de ajuste (cast), transformando os inteiros em string. O método array(listas, dtype) aceita o parâmetro dtype onde se pode informar o tipo de elemento que se pretende armazenar.
» stArr = np.array([['a', 'b'],['c', 'd']])
» stArr
↳ array([['a', 'b'],
['c', 'd']], dtype='<U1')
» stArr[0,1]
↳ 'b'
» stArr.dtype
↳ dtype('<U1')
» stArr.dtype.name
↳ 'str32'
» stArr2 = np.array([[1.01, 2.02],['h', 'i']])
» stArr2
↳ array([['1.01', '2.02'],
['h', 'i']], dtype='<U21')
» stArr3 = np.array([[True, False],['h', 'i']])
» stArr3
↳ array([['True', 'False'],
['h', 'i']], dtype='<U5')
» # parâmetro dtype
» cplx = np.array([[1, 2, 3],[4, 5, 6]], dtype=complex)
» cplx
↳ array([[1.+0.j, 2.+0.j, 3.+0.j],
[4.+0.j, 5.+0.j, 6.+0.j]])
Arrays podem ser transformados de um tipo para outro (quando possível). Para isso usamos array.astype(). Na transformação de floats para inteiros a parte decimal será truncada. Arrays de strings, desde que devidamente formatados, podem ser convertidos em numéricos.
» # criando um array de integers
» arr = np.array([1, 2, 3, 4, 5])
» arr.dtype
↳ dtype('int64')
» # cast para array de ponto flutuante
» floatArr = arr.astype(np.float64)
» floatArr.dtype
↳ dtype('float64')
» # floats para integers
» # criando um array de floats
» arr = np.array([1.9, -8.2, -9.6, 0.9, 2.3, 10.7])
» arr
↳ array([ 1.9, -8.2, -9.6, 0.9, 2.3, 10.7])
» # converte para inteiros (trunca parte inteira)
» arr.astype(np.int32)
↳ array([ 1, -8, -9, 0, 2, 10], dtype=int32)
» # um array de strings
» arrNumStrings = np.array(['0.0', '7.75', '-6.6', '100'], dtype=np.string_)
» arrNumStrings.astype(float)
↳ array([ 0., 7.75, -6.6 , 100.]
Métodos predefinidos de construção
O método np.arange(m,n,[p]) retorna um array de inteiros no intervalo (m, n], i.e., começando em m e terminando em n-1. Se o primeiro argumento for omitido m=0. Um terceiro argumento informa o p=passo, intervalo entre os valores da sequência. Em np.arange(m,n,p) m, n devem ser inteiros mas p pode ser um float.
O método np.linspace(m,n,p) retorna um array no intervalo (m, n), ambos os extremos incluídos, com p números igualmente espaçados.
np.random.random(n) retorna um array com n elementos aleatórios e np.random(m, n) retorna um array com shape = (m,n) e elementos aleatórios.
» # np.range(n)
» np.arange(10)
↳ array([0, 1, 2, 3, 4, 5, 6, 7, 8, 9])
» # np.range(m,n)
» np.arange(5, 15)
↳ array([ 5, 6, 7, 8, 9, 10, 11, 12, 13, 14])
» # np.range(m,n,p)
» np.arange(10, 100, 10)
↳ array([10, 20, 30, 40, 50, 60, 70, 80, 90])
» np.arange(10, 20, .5)
↳ array([10. , 10.5, 11. , 11.5, 12. , 12.5, 13. , 13.5, 14. , 14.5, 15. ,
15.5, 16. , 16.5, 17. , 17.5, 18. , 18.5, 19. , 19.5])
» # np.linspace(m,n,p)
» np.linspace(10, 20, 5)
↳ array([10. , 12.5, 15. , 17.5, 20. ])
» # a sequência pode ser decrescente
» np.linspace(20, 10, 5)
↳ array([20. , 17.5, 15. , 12.5, 10. ])
» # np.random.random(n)
» np.random.random(10)
↳ array([0.35433322, 0.54555179, 0.48783323, 0.5785414 , 0.76837232,
0.69888297, 0.62492788, 0.33289321, 0.75068313, 0.95667854])
» # np.random.random(m,n)
» np.random.random((2,3))
↳ array([[0.11351481, 0.76831577, 0.27597676],
[0.73130126, 0.7225559 , 0.54040225]])
Os métodos np.zeros((m,n)) e np.ones((m,n)) criam, respectivamente, ndarrays de zeros e uns com as dimensões dadas pela tupla (ou lista) no argumento. np.eye(m,n) gera um ndarray m × n com elementos 1 na diagonal, 0 fora dela. np.eye(n,n) é o mesmo que np.eye(n) ou np.identity(n), que são a matriz identidade de n dimensões.
» np.zeros((2,4))
↳ array([[0., 0., 0., 0.],
[0., 0., 0., 0.]])
» np.ones((2,3))
↳ array([[1., 1., 1.],
[1., 1., 1.]])
» np.eye(2,4)
↳ array([[1., 0., 0., 0.],
[0., 1., 0., 0.]])
» np.identity(3)
↳ array([[1., 0., 0.],
[0., 1., 0.],
[0., 0., 1.]])
» np.eye(2,6)
↳ array([[1., 0., 0., 0., 0., 0.],
[0., 1., 0., 0., 0., 0.]])
» np.eye(4,4)
↳ array([[1., 0., 0., 0.],
[0., 1., 0., 0.],
[0., 0., 1., 0.],
[0., 0., 0., 1.]])
O método np.empty(m,n) permite a criação de arrays vazios, em geral destinados a serem preenchidos depois de sua criação por meio de algum cálculo ou leitura de dados. Nos exemplos criamos um array numérico vazio, arrN. Observe que não há garantia de que as entradas serão nulas. Em seguida criamos um array vazio de strings, com espaço para 3 caracteres (dtype='<U3′), e o preenchemos em um loop.
» # array numérico "vazio"
» arrN = np.empty((1,3))
» arrN
↳ array([[4.66896202e-310, 0.00000000e+000, 1.58101007e-322]])
» # array de strings
» arr = np.empty((3,3), dtype='<U3')
» arr
↳ array([['', '', ''],
['', '', ''],
['', '', '']], dtype='<U3')
» for linha in range(arr.shape[0]):
» for coluna in range(arr.shape[1]):
» arr[linha,coluna] = 'a' + str(linha) + str(coluna)
» arr
↳ array([['a00', 'a01', 'a02'],
['a10', 'a11', 'a12'],
['a20', 'a21', 'a22']], dtype='<U3')
Alterando dimensões
Dado um array de uma única linha com r elementos podemos tranformá-lo em um array com shape = (m,n), desde que as dimensões sejam compatíveis, i.e., r = m × n. De fato, qualquer array pode ser transformado em outro se eles possuem o mesmo número de elementos (mesmo size ).
» arr = np.random.random(12)
» arr
↳ array([0.04276829, 0.76468762, 0.24807651, 0.75531679, 0.60327475,
0.81704922, 0.08233836, 0.64112484, 0.55276595, 0.30669723,
0.43989324, 0.60031761])
» # reshape
» arr.reshape(3,4)
↳ array([[0.04276829, 0.76468762, 0.24807651, 0.75531679],
[0.60327475, 0.81704922, 0.08233836, 0.64112484],
[0.55276595, 0.30669723, 0.43989324, 0.60031761]])
» np.linspace(0,10, 6).reshape(2,3)
↳ array([[ 0., 2., 4.],
[ 6., 8., 10.]])
Indexação e fatiamento

Quando um array é criado ele recebe automaticamente um conjunto de índices. Um elemento pode ser lido ou alterado por meio de seu índice. Índices negativos contam de trás para frente, sendo arr[-1] o último elemento do array. Para selecionar (ou editar) vários elementos passamos uma lista de índices.
» arr = np.linspace(1,12, 12)
» arr
↳ array([ 1., 2., 3., 4., 5., 6., 7., 8., 9., 10., 11., 12.])
» # o 5º elemento
» arr[4]
↳ 5.
» # alterando o 5º elemento
» arr[4] = 100
» arr
↳ array([ 1., 2., 3., 4., 100., 6., 7., 8., 9., 10., 11., 12.])
» arr[-8]
↳ 100
» # lendo vários elementos
» arr[[0,2,5]]
↳ array([1., 3., 6.])
» # alterando vários elementos
» arr[[0,2,5]] = [10, 20, 30]
» arr
↳ array([ 10., 2., 20., 4., 100., 30., 7., 8., 9., 10., 11., 12.])
 No caso de arrays bidimensionais os elementos do array são acessados pelos índices de suas linhas e colunas, sendo que
No caso de arrays bidimensionais os elementos do array são acessados pelos índices de suas linhas e colunas, sendo que arr[l,c] = al,c é o elemento da linha l e coluna c. Em objetos de ranks superiores cada índice se refere a um dos eixos.
Uma fatia ou slice do array é um subconjunto de elementos que pode ter o mesmo shape ou não. Para um vetor, digamos arr1D = [a0, a1, ..., aM] a fatia arr1D[m,n] = [am, ..., an-1], onde m ≥ 0, n ≤ M. Vale lembrar que o comprimento da fatia de um array unidimensional é arr1D[m,n].size = n-m.
» # outro teste para slices
» arr = np.arange(10) # cria o array array([0, 1, 2, 3, 4, 5, 6, 7, 8, 9])
» arr[6:9]
↳ array([6, 7, 8])
» arr[:3]
↳ array([0, 1, 2])
» arr[7:]
↳ array([7, 8, 9])
» # uma seção pode ser alterada†
» arr[3:6] = -5
» arr
↳ array([ 0, 1, 2, -5, -5, -5, 6, 7, 8, 9])
» arr[7:] = -arr[7:]
» arr
↳ array([ 0, 1, 2, -5, -5, -5, 6, -7, -8, -9])
(†) Uma operação entre arrays de dimensões diferentes, como ocorre em arr[0:4] = -1 é chamado de propagação ou broadcasting. Para interagir com a 1ª parte da expressão a 2ª é transformada: -1 → arr[-1,-1,-1,-1]. Voltaremos a esse tópico.
Observe que uma fatia de um array é uma referência àquela parte do array e qualquer alteração feita na fatia se refletirá no array original. A notação arr[i:j] significa, claro, elementos de i-ésimo até (j-1)-ésimo. arr[:] significa todos os elementos do array.
» # criando um array de teste
» arr = (np.random.random(10)*10).round(1)
» arr
↳ array([3. , 1.5, 5.3, 1.3, 8.8, 9.8, 4.7, 0.1, 0.1, 0.6])
» fatia = arr[1:5]
» fatia
↳ array([1.5, 5.3, 1.3, 8.8])
# vamos alterar trecho da fatia
» fatia[1:3] = 0
» fatia
↳ array([1.5, 0. , 0. , 8.8])
» # o array original foi alterado
» arr
↳ array([3. , 1.5, 0. , 0. , 8.8, 9.8, 4.7, 0.1, 0.1, 0.6])
» # vamos alterar a fatia inteira
» fatia[:] = -10
» arr
↳ array([ 3. , -10. , -10. , -10. , -10. , 9.8, 4.7, 0.1, 0.1, 0.6])
» # valores específicos podem ser fornecidos (sem broadcast)
» fatia[:] = [-1,-2,-3,-4]
» arr
↳ array([ 3. , -1. , -2. , -3. , -4. , 9.8, 4.7, 0.1, 0.1, 0.6])
Esse comportamento é útil quando se trabalha com array de dados muito grande e se deseja alterar apenas parte dele, lembrando que a biblioteca efetua suas operações mantendo os dados envolvidos na memória.
Em um array arr de 2 dimensões arr[m] é a m-ésima linha e arr[m,n] se refere ao elemento am,n, da m-ésima linha, n-ésima coluna. arr[m][n] é o mesmo am,n.
» # slices para dimensões mais altas
» arr = np.array([[1, 2, 3], [4, 5, 6], [7, 8, 9]])
» arr
↳ array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
» # 2ª linha
» arr[1]
↳ array([4, 5, 6])
» arr[:1]
↳ array([[1, 2, 3]])
» # 1ª linha, 3º elemento
» arr[0,2]
↳ 3
» arr[0][2]
↳ 3
Uma cópia de um setor é uma referência para aquele setor, chamda de view ou visualização do segmento. Alterações feitas à view se refletam no array original, a menos que o método arr.copy() seja usado. Um array copiado dessa forma perde a referência com o array original e pode ser modificado independentemente.
» # slices em 3D:
» # criamos um array com shape (2,3,2)
» arr3D = np.arange(12).reshape(2,3,2)
» arr3D
↳ array([[[ 0, 1],
[ 2, 3],
[ 4, 5]],
[[ 6, 7],
[ 8, 9],
[10, 11]]])
» # a 1ª matriz é
» arr3D[0]
↳ array([[0, 1],
[2, 3],
[4, 5]])
» # a 2ª linha da 1ª matriz é
» arr3D[0,1]
↳ array([2, 3])
» # seu 2º elemento
» arr3D[0,1,1] # o mesmo que arr3D[0,1][1]
↳ 1
» # copiamos um slice de 2 formas
» guardar = arr3D[0].copy()
» slice = arr3D[0]
» # ambos com os valores da 1ª matriz
» # alteramos toda a 1ª matriz
» arr3D[0] = 12
» # o array original foi alterado
» arr3D
↳ array([[[12, 12],
[12, 12],
[12, 12]],
[[ 6, 7],
[ 8, 9],
[10, 11]]])
» # a cópia por referência foi alterada
» slice
↳ array([[12, 12],
[12, 12],
[12, 12]])
» # mas a cópia por valor não foi alterada
» guardar
↳ array([[0, 1],
[2, 3],
[4, 5]])
» # podemos restaurar o array aos seus valores originais
» arr3D[0] = guardar
» arr3D
↳ array([[[ 0, 1],
[ 2, 3],
[ 4, 5]],
[[ 6, 7],
[ 8, 9],
[10, 11]]])
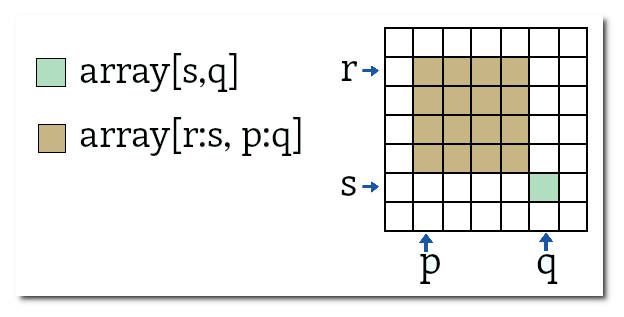
A notação de slice, idêntica à usada em listas do python, funciona em arrays. Para um array unidimensional arr1d[i:f] retorna do i-ésimo elemento até j-ésimo elemento (exclusive). Para um array de 2 dimensões arr2d[i:f] retorna i-ésima linha até j-ésima linha (exclusive). Se i é omitido o início é usado, se j é omitido o final é usado. Portanto arr2d[:2] significa as duas primeiras linhas do array (linha 0 e linha 1).
Slices ou segmentos múltiplos podem ser usados. Por exemplo, arr2d[m:n, r:s] são as linhas de m até n-1, colunas de r até s-1.
» # array 1d (um vetor)
» arr = np.array([1.2, 2.3, 3.4, 4.5, 5.6])
» arr[2:4]
↳ array([3.4, 4.5])
» # array 2d (uma matriz)
» arr2d = np.array([[1,2,3],[4,5,6],[7,8,9]])
» arr2d
↳ array([[1, 2, 3],
[4, 5, 6],
[7, 8, 9]])
» arr2d[0:2]
↳ array([[1, 2, 3],
[4, 5, 6]])
» arr2d[:2]
↳ array([[1, 2, 3],
[4, 5, 6]])
» # slices múltiplos
» arr2d[:2, 1:]
↳ array([[2, 3],
[5, 6]])
arr2d[:2, 1:] são as linhas 0 e 1, colunas de 1 em diante.
O indexador pode ser um array booleano, e os valores serão filtrados apenas se o índice for True. Essa operação pode ser muito útil para a implementação de filtragens de tipos diversos.
» # o indexador pode ser booleano
» ar1 = np.array([-1,0, 3, 7, -2, 10])
» ar2 = np.array([-2,10, 2, 9, -1, 8])
» # extrair apenas 0º e 2º elemento
» ar1[[True, False, True, False, False, False]]
↳ array([-1, 3])
» # elementos de ar1 maiores que 2
» ar1[ar1>2]
↳ array([ 3, 7, 10])
» # elementos de ar1 maiores que os de ar2
» ar1[ar1>ar2]
↳ array([-1, 3, 10])

Suponha que temos dados sobre alguns países, armazenados em forma tabular. O primeiro array contém os nomes dos países, como se fosse um cabeçalho da tabela seguinte. O segundo array contém dados numéricos de qualquer natureza, com 5 linhas e 4 colunas, cada coluna contendo dados relativos ao país no cabeçalho. Para esse exemplo geramos esses dados aleatoriamente, apenas para exibir a operação.
» arrPais = np.array(['Brasil', 'Chile', 'Brasil', 'Peru'])
» arrDados = np.random.randn(5,4).round(2) # 5 linhas e 4 colunas
» print(arrPais, '\n', arrDados)
↳ ['Brasil' 'Chile' 'Brasil' 'Peru']
[[-0.71 0.42 -0.52 0.63]
[-1.12 -0.29 0.03 1.43]
[ 0.99 0.45 1.08 0.53]
[-0.78 0.18 -0.07 0.28]
[-2.03 0.44 0.07 1.28]]
» # podemos exibir todas as linhas das colunas 0 e 2
» arrDados[:,[0,2]]
↳ array([[-0.71, -0.52],
[-1.12, 0.03],
[ 0.99, 1.08],
[-0.78, -0.07],
[-2.03, 0.07]])
» # alternativamente, as colunas que correspondem ao Brasil
» arrBrasil = arrDados[:, arrPais=='Brasil']
» arrBrasil
↳ array([[-0.71, -0.52],
[-1.12, 0.03],
[ 0.99, 1.08],
[-0.78, -0.07],
[-2.03, 0.07]])
» # na tabela do Brasil, suponha que valores negativos sejam insignificantes
» # podemos eliminá-los com uma filtragem
» arrBrasil[arrBrasil < 0] = 0
» arrBrasil
↳ array([[0. , 0. ],
[0. , 0.03],
[0.99, 1.08],
[0. , 0. ],
[0. , 0.07]])
» # a soma desses dados é
» arrBrasil.sum()
↳ 2.17
» # para exibir os demais dados (não do Brasil)
» arrDados[:, arrPais!='Brasil'] # ou arrDados[:, ~(arrPais=='Brasil')]
↳ array([[ 0.42, 0.63],
[-0.29, 1.43],
[ 0.45, 0.53],
[ 0.18, 0.28],
[ 0.44, 1.28]])
Observe que arrPais!='Brasil' é a negação de arrPais=='Brasil'. O parênteses em ~(arrPais=='Brasil') é necessário pois a negação ~ tem precedência sobre o teste de igualdade. Sem o parênteses ~arrPais seria avaliado primeiro, o que resultaria em erro pois o array não é booleano.
As ferramentas do pandas facilitam operações como essas.
Operações matemáticas
Operações são realizadas elemento a elemento (que abreviaremos para “e/e” nesse texto). Operações usuais de um array por um escalar são são propagadas entre o escalar e cada elemento do array. Operações entre arrays são feitas e/e, ou seja, realizadas entre elementos na mesma posição. Os arrays devem ter as mesmas dimensões.
Operações de incremento, tais como array += 1 (que significa array = array + 1) ou array *= 2 são realizados inplace (alteram o próprio array). Diversas outras operações do NumPy (e do pandas) são realizadas inplace enquanto em várias delas existe o parâmetro inplace = True/False que permite a decisão de qual caso se deseja naquele momento.
Operações podem ser feitas entre arrays retornados por funções (que retornam arrays).
» # operações com escalares
» ar1 = np.linspace(10, 20, 5)
» ar1
↳ [10. 12.5 15. 17.5 20. ]
» ar1 + 10
↳ [20. 22.5 25. 27.5 30. ]
» ar1 * 10
↳ [100. 125. 150. 175. 200.]
» ar1 / 10
↳ [1. 1.25 1.5 1.75 2. ]
» ar1**2
↳ [100. 156.25 225. 306.25 400. ]
» 1/arr1
↳ array([0.1 , 0.08 , 0.06666667, 0.05714286, 0.05 ])
» # operações entre arrays
» ar2 = np.linspace(10, 50, 5)
» print(ar1)
» print(ar2)
↳ [10. 12.5 15. 17.5 20. ]
↳ [10. 20. 30. 40. 50.]
» ar1 + ar2
↳ array([20. , 32.5, 45. , 57.5, 70. ])
» ar1 * ar2
↳ array([ 100., 250., 450., 700., 1000.])
» ar2 / ar1
↳ array([1. , 1.6 , 2. , 2.28571429, 2.5 ])
» # operações com arrays (2 × 3)
» ar3 = np.linspace(0,5, 6).reshape(2,3)
» ar4 = np.linspace(0,10, 6).reshape(2,3)
» print(ar3)
↳ [[0. 1. 2.]
[3. 4. 5.]]
» print(ar4)
↳ [[ 0. 2. 4.]
[ 6. 8. 10.]]
» ar3 + ar4
↳ array([[ 0., 3., 6.],
[ 9., 12., 15.]])
» ar3 - ar4
↳ array([[ 0., -1., -2.],
[-3., -4., -5.]])
» ar3 * ar4
↳ array([[ 0., 2., 8.],
[18., 32., 50.]])
» # operações de incremento são realizados inplace
» ar3 +=1
» ar3
↳ array([[1., 2., 3.],
[4., 5., 6.]])
» # seno e cosseno em np retorna um array e/e
» ar1 * np.sin(ar2)
↳ array([ -5.44021111, 11.41181563, -14.82047436, 13.03948031,
-5.24749707])
» ar3 * np.cos(ar4)
↳ array([[ 0. , -0.41614684, -1.30728724],
[ 2.88051086, -0.58200014, -4.19535765]])
Comparações entre arrays resultam em arrays booleanos.
» ar5 = np.linspace(0,5, 6).reshape(2,3)
» ar6 = np.random.random(6).reshape(2,3).round(2)*5
» ar5
↳ array([[0., 1., 2.],
[3., 4., 5.]])
» ar6
↳ array([[3. , 2.45, 1.2 ],
[3.1 , 1.55, 1.75]])
» arrMaior= ar5 > ar6
» arrMaior
↳ array([[False, False, True],
[False, True, True]])
Broadcasting: A operações feitas acima, entre um array e um escalar, transformam o escalar em um array de dimensões apropriadas (de mesmo shape) antes de sua realização. Essa operação se chama broadcasting:. Por exemplo:
» ar1 = np.array([0,1,2,3])
» ar2 = np.array([4,4,4,4])
» # a soma com um escalar
» ar1 + 4
↳ array([4, 5, 6, 7])
» # é mesmo que
» ar1 + ar2
↳ array([4, 5, 6, 7])
» # os elementos são iguais
» ar1 + ar2 == ar1 + 4
↳ array([ True, True, True, True])
# o mesmo ocorre com comparações
» ar1 ≥ 2
↳ array([False, False, True, True])
Uma forma de seleção diferente consiste em passar listas de valores como índices. O array retornado depende de como essas listas são passadas. Essa é técnica é chamada de fancy indexing (indexação sofisticada). Em qualquer dos casos abaixo os índices podem aparecer em qualquer ordem.
O slice array[[i,j,...]] contém as linhas array[i], array[j], etc. (O mesmo que array[:,[i,j,...]]).
O slice array[:,[i,j,...]] contém as colunas array[:,i], array[:,j], etc.
O slice array[[i,j,...]:[r, s,...]] é uma linha contendo os elementos array[i,r], array[j,s], etc.
» # fancy indexing (passando arrays como indices)
» # vamos construir um array 6 × 5 e atribuir seus valores um a um
» arr = np.empty((6,5))
» for linha in range(6):
» for coluna in range(5):
» arr[linha,coluna] = linha * 10 + coluna
» # o array obtido é (uma forma de identificar facilmente de que elemento se trata)
» arr
↳ array([[ 0., 1., 2., 3., 4.],
[10., 11., 12., 13., 14.],
[20., 21., 22., 23., 24.],
[30., 31., 32., 33., 34.],
[40., 41., 42., 43., 44.],
[50., 51., 52., 53., 54.]])
» # podemos selecionar linhas (em qualquer ordem)
» arr[[3,5,1]] # o mesmo que arr[[3,5,1],:]
↳ array([[30., 31., 32., 33., 34.],
[50., 51., 52., 53., 54.],
[10., 11., 12., 13., 14.]])
» # ou colunas (em qualquer ordem)
» arr[:,[3,1]]
↳ array([[ 3., 1.],
[13., 11.],
[23., 21.],
[33., 31.],
[43., 41.],
[53., 51.]])
» # fornecer duas listas (que devem ter o mesmo tamanho) tem efeito diferente,
» # retornando array de i dimensão com os índices dados nas listas
» arr[[0,3,4,1],[0,3,4,1]]
↳ array([ 0., 33., 44., 11.])
» arr[[0,3,4,1],[1,0,4,2]]
↳ array([ 1., 30., 44., 12.])
Funções universais
Funções universais, ou ufunc, são funções que agem e/e, sobre todos os elementos de um array, retornando outra array de mesmas dimensões. Essas operações são também chamadas de operações vetorializadas. Embora envolvam laços (loops ) esses são realizados internamente e de forma eficiente, de modo a agilizar os processos.
Uma tabela das funções universais é encontrada abaixo.
| Método |
retorna |
abs, fabs |
valor absoluto inteiros, floats, ou complexos |
sqrt |
raiz quadrada (equivale a arr**0.5) |
square |
elementos elevados ao quadrado (equivale a arr**2) |
exp |
exponencial de cada elemento (ex) |
log, log10, |
logaritmos naturais (de base e e base 10var> |
log2, log1p |
logaritmos de base 2 e log(1 + x) |
sign |
sinal: (1, 0, -1) (positivo, zero, negativo) |
ceil |
teto, menor inteiro maior ou igual |
floor |
piso, maior inteiro menor ou igual |
rint |
arredonda para o inteiro mais próximo, preservando dtype |
modf |
partes inteiras e fracionárias do array, em e arrays |
isnan |
array booleano, se o valor é NaN (Not a Number) |
isfinite |
array booleano, se cada valor é finito (non-inf, non-NaN) |
isinf |
array booleano, se cada valor é infinito |
cos, sin, tan |
funções trigonométricas |
cosh, sinh, tanh |
funções trigonométricas hiperbólicas |
arccos, arcsin, arctan |
arcos de funções trigonométricas |
arccosh, arcsinh, arctanh |
arcos de funções trigonométricas hiperbólicas |
logical_not |
array booleano, negação do array (equivalent to ~arr). |
Exemplos de uso:
» # Funções universais
» arrBool = np.array([True, False, False, True])
» arrBool
↳ array([ True, False, False, True])
» np.logical_not(arrBool)
↳ array([False, True, True, False])
» arr = np.linspace(0, 10, 6)
» arr -=5
» arr
↳ array([-5., -3., -1., 1., 3., 5.])
» np.abs(arr)
↳ array([5., 3., 1., 1., 3., 5.])
» # não altera arr
» np.sign(arr)
↳ array([-1., -1., -1., 1., 1., 1.])
» arr = arr/10 +4
» arr = arr.reshape(2,3)
» arr
↳ array([[3.5, 3.7, 3.9],
[4.1, 4.3, 4.5]])
» np.modf(arr)
↳ (array([[0.5, 0.7, 0.9],
[0.1, 0.3, 0.5]]),
↳ array([[3., 3., 3.],
[4., 4., 4.]]))
Funções de Agregação

Funções de agregação são funções que realizam operações em todos os elementos do array, retornando um escalar(um número). Em sua maioria elas retornam cálculos estatíscos sobre os dados.
» ag = np.array([3.3, 12.5, 11.2, 5.7, 0.3])
» ag
↳ array([ 3.3, 12.5, 11.2, 5.7, 0.3])
» # outputs nos comentários
» ag.sum() # 33.0
» ag.min() # 0.3
» ag.max() # 12.5
» ag.mean() # 6.6
» ag.std() # 4.6337889464238655
» ag.var() # 21.472
» ag.argmin() # 4
» ag.argmax() # 1
» # o mesmo vale para arrays com outros shapes
» ag23 = np.random.random(6).reshape(2,3) -.5
» ag23
↳ array([[ 0.10425075, -0.29335437, -0.36814244],
[ 0.32986805, 0.17289794, -0.4568041 ]])
» ag23.mean()
↳ -0.08521402850598175
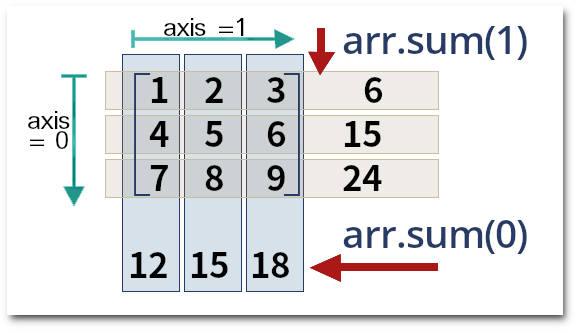
Diversas das operações podem ser feitas sobre o array inteiro ou sobre linhas ou colunas. Para isso podemos especificar axis = 0 para operações sobre elementos das colunas, axis = 1 para operações sobre elementos das linhas.
» arr = np.random.randn(4,5).round(2)
» arr
↳ array([[ 0.45, -0.11, -0.54, -0.97, -0.23],
[ 1.65, 0.76, -0.39, -1.83, 0.02],
[ 0.45, -1.22, 1.93, 1.92, -0.43],
[-0.39, 2.01, 0.04, 0.67, -1.1 ]])
» # a soma de todos os elementos
» arr.sum()
↳ 2.689999999999999
» # soma sobre elementos de cada coluna
» arr.sum(axis=0) # ou arr.sum(0)
↳ array([ 2.17, 0.37, 0.17, -2.49, -3.13])
» # soma sobre elementos de cada linha
» arr.sum(axis=1) # ou arr.sum(1)
↳ array([-0.42, -1.67, 0.6 , -1.42])
» # produtos dos elementos das linhas
» arr.prod(axis=1).round(2)
↳ array([0.01, 0.02, 0.87, 0.02])
» # produtos dos elementos das colunas
» arr.prod(axis=0).round(2)
↳ array([-0.13, 0.21, 0.02, 2.28, -0. ])
Funções básicas de agregação:
| Função |
retorna |
np.all |
booleano, True se todos os elementos no array são não nulos |
np.any |
booleano, True se algum dos elementos no array é não nulo |
np.sum |
soma dos elementos do array ou sobre eixo especificado †. |
np.mean |
Média aritmética; arrays de comprimento nulo têm média = NaN |
np.std, np.var |
variância e desvio padrão |
np.max, np.min |
valor máximo e mínimo no array |
np.argmax, np.argmin |
índices do valor máximo e mínimo no array |
np.cumsum |
soma cumulativa dos elementos, começando em 0 |
np.cumprod |
produto cumulativo dos elementos, começando em 1 |
(†) Para arrays de comprimento nulo tem soma np.sum = NaN.
Bibliografia
- Blair, Steve: Python Data Science, edição do autor, 2019.
- Harrison, Matt: Learning Pandas, Python Tools for Data Munging, Data Analysis, and Visualization,
Treading on Python Series, Prentiss, 2016.
- Johansson, Robert: Numerical Python, Scientific Computing and Data Science Applications with Numpy, SciPy and Matplotlib, 2nd., Chiba, Japan, 2019.
- McKinney, Wes: Python for Data Analysis, O’Reilly Media, Sebastopol CA, 2018.
- McKinney, Wes, Pandas Development Team: pandas: powerful Python data analysis toolkit Release 1.2.1,
- Miller, Curtis: Hands-On Data Analysis with NumPy and pandas, Packt Publishing, Birmingham, 2018.
- Nelli, Fabio: Python Data Analytics With Pandas, NumPy, and Matplotlib, 2nd., Springer, New York, 2018.
- Site AI Ensina: Entendendo a biblioteca NumPy, acessado em julho de 2021.
- Site GeeksforGeeks: Python NumPy, acessado em julho de 2021.
- Site W3 Schools: NumPy Tutorial, acessado em julho de 2021.
- NumPy, docs.
- NumPy, Learn.
Nesse site:










 Leia:
Leia:















 » # A A-1 é a identidade
» np.dot(arr,inv(arr))
↳ array([[1., 0., 0.],
[0., 1., 0.],
[0., 0., 1.]])
» # A A-1 é a identidade
» np.dot(arr,inv(arr))
↳ array([[1., 0., 0.],
[0., 1., 0.],
[0., 0., 1.]])







 No caso de arrays bidimensionais os elementos do array são acessados pelos índices de suas linhas e colunas, sendo que
No caso de arrays bidimensionais os elementos do array são acessados pelos índices de suas linhas e colunas, sendo que